Leibniz-Institut DSMZ GmbH
Biologische Charakterisierung und Auswahl therapietauglicher Phagen
Damit ein Patient, welcher mit einem multiresistenten Bakterium (bspw. Staphylococcus aureus in Form von MRSA oder 3/4MRGN Pseudomonas aeruginosa) besiedelt/infiziert ist, mit Bakteriophagen (kurz: Phagen) behandelt werden kann, müssen zunächst gut geeignete lytische Phagen identifiziert und bereitgestellt werden. Ein gut geeigneter Phage zeichnet sich dadurch aus, dass er zum einen möglichst viele Bakterienvarianten („Stämme“, „Isolate“) einer Art zerstören kann und zum anderen als unbedenklich eingestuft ist. Dazu werden Phagen aus der Sammlung des Leibniz-Instituts DSMZ GmbH auf einer diversen Sammlung von Patientenisolaten getestet. Zudem müssen diese Phagen genetisch untersucht werden, damit ausgeschlossen werden kann, dass sie Virulenzfaktoren oder Antibiotikaresistenzmarker tragen. Vielversprechende Kandidaten werden nachfolgend in vitro (im Labor) auf die Effizienz Bakterien zu zerstören erforscht. Gleichzeitig erfolgt die Suche nach weiteren neuen Phagen in unterschiedlichen Umweltproben, um das Repertoire zu vergrößern. Es folgt die klonale Vereinzelung/klonale Reinigung der Phagen, Vermehrung in kleinem Maßstab und ebenfalls die Untersuchung auf ihre Eignung für Anwendungszwecke. Da jeder Phage innerhalb einer Bakterienart nur bestimmte Patientenisolate zerstören kann, ist es notwendig verschiedene Phagen zu finden, die gemeinsam möglichst viele Bakterienisolate abdecken. Für jede im Projekt PhagoFlow priorisierte Bakterienart soll daher ein Set an Phagen mit hoher genetischer und morphologischer Diversität zusammengestellt werden, so erhöht sich die Wahrscheinlichkeit ausreichender Abdeckungen von Patientenisolaten.
Im Zuge des Projekts PhagoFlow soll rechtzeitig das Verfahren eines Phagogramms für die Kliniklaborroutine zur Empfindlichkeitsprüfung von Patientenisolaten gegenüber Phagen entwickelt werden. Dazu ist es notwendig verschiedene Methoden miteinander auf Durchführbarkeit, Auswertbarkeit und Reproduzierbarkeit zu vergleichen. Das Ziel ist eine einfache Durchführung und Harmonisierung der Inkubationsbedingungen, damit dieses Verfahren gut in den Routine-Alltag der Diagnostik integriert werden kann.
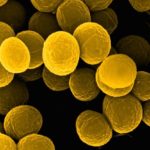
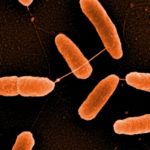
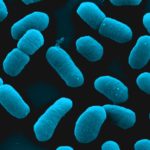
Zunächst werden für drei Bakterienarten aus der ESKAPE-Gruppe effiziente neue Phagen gesucht: 1.) Staphylococcus aureus (mit sehr hoher Häufigkeit als Infektionserreger im BWkrhs festgestellt) ist ein kugelförmiges gram-positives Bakterium, ein normaler Bewohner der Haut und des Nasen-Rachenraums. Stämme dieser Bakterienart sind jedoch oft befähigt, verschiedene Toxine zu produzieren, ferner ist das Bakterium als Eitererreger bei Entzündungsprozessen bekannt. Gerät das Bakterium in die Blutbahn, so können gravierende Folgen entstehen, es ist einer der häufigsten bekannten Erreger der Bakteriämie und Sepsis, siedelt sich aber auch an Organen wie z. B. an Herzklappen sowie an körperfremden Oberflächen ab wie z. B. an künstlichen Herzklappen oder Gelenkersatz. Bei S. aureus sind die MRSA-Varianten, Methicillin-resistente Stämme, besonders gefürchtet, der sprichwörtliche sogenannte „Krankenhauskeim“, dessen antibiotische Behandlung schwierig ist. 2.) Ebenso ist Pseudomonas aeruginosa ein im BWkrhs sehr häufig festgestellter Infektionserreger, ein gram-negatives Stäbchenbakterium, das quasi überall in der Umwelt und in und auf uns vorkommt, ein überaus flexibles Genom hat und aufgrund dieser Flexibilität der genetischen Ausstattung sehr anpassungsfähig ist. Besonders typisch kommt P. aeruginosa in Biofilmen vor, mikrobielle Lebensgemeinschaften, in denen die Bakterien zwar „ruhen“, aber intensiven Austausch von Genen und Information pflegen (s. auch „Quorum sensing“), bereit, auch Virulenzfaktoren und Antibiotikaresistenzen zum gegenseitigen Selektionsvorteil auszutauschen. Die genannten unerwünschten Eigenschaften von S. aureus treffen fast vollumfänglich auch für P. aeruginosa zu, wobei P. aeruginosa allgemein bei verschiedenen Infektionen noch etwas häufiger ist. Bei dieser Bakterienart sind mittlerweile nicht nur multi- sondern sogar pan-resistente Stämme bekannt, bei denen kein einziges therapietaugliches Antibiotikum mehr anschlägt. 3.) Mit mäßiger Häufigkeit wird im BWkrhs das ebenfalls gram-negative Stäbchen Escherichia coli diagnostiziert, ein typischer Darmbewohner, der innerhalb seiner Art vom Status des wichtigen Mikrobiombewohners bis hin zum lebensgefährlichen Pathotyp EHEC eine gewaltige Breite an Varianten aufweist, mit Stämmen, die weder Toxine bilden noch Virulenzfaktoren tragen bis zu extrem gefährlichen Toxinbildnern mit raffinierten Infektionsmechanismen und Antibiotika-Multiresistenzen. Die Bedeutung der Feindiagnostik im Falle von E. coli bei Patienten ist daher besonders deutlich. Diese drei Bakterienarten haben zudem gemeinsam dass sie sich sehr schnell vermehren. Auch in Mischinfektionen kommen sie vor. Im weiteren Projektverlauf können nach Bedarf und Möglichkeiten weitere Bakterienarten in den Fokus rücken.